Точна репарація дволанцюгового розриву ДНК можлива лише підчас реплікації, коли розірвана ділянка може бути відновлена завдяки використанню сестринської молекули як матриці за механізмом гомологічної рекомбінації. В інших випадках цілісність розірваних молекул ДНК відновлюється в консервативному для всіх організмів процесі негомологічного з’єднання кінців (NHEJ – Non Homologous End Joining). З’єднуються при цьому будь-які кінці ДНК, що, за наявності великої кількості розривів, призводить до об’єднання ділянок різних хромосом, транслокацій тощо.
Ключову роль у процесі NHEJ відіграють білки Ku, які (у гетеродимерній формі) упізнають кінці ДНК, об’єднують їх у не ковалентний комплекс і рекрутують до цього комплексу низку інших білків.У складі комплексу відбувається розплітання кінцевих ділянок ДНК і пошук мікрогомології – коротких комплементарних (чи майже комплементарних) одноланцюгових ділянок, які належать різним молекулам (рис. 28). У результаті утворюється короткий дуплекс, зайві одноланцюгові ділянки відщеплюються нуклеазами, лігаза зшиває одноланцюгові розриви. Таким чином, у процесі з’єднання відбувається втрата кількох пар основ. Ця особливість NHEJ-системи використовується при рекомбінації імуноглобулінових генів з метою підвищення розмаїття їхніх послідовностей.
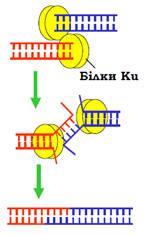
Рис. 28. Негомологічне з’єднання кінців ДНК.
КОНТРОЛЬНІ ЗАПИТАННЯ
1. Що таке реплікон? Охарактеризуйте основні типи репліконів.
2. Дайте визначення реплікативної вилки. Яка різниця між двома ланцюгами ДНК, що синтезуються під час реплікації? Що таке фрагменти Оказакі?
3. Які ферментативні активності мають бактеріальні ДНК-полімерази? Порівняйте особливості й функціональне значення ДНК-полімераз І і ІІІ.4. Опишіть основні риси просторової структури ДНК-полімераз.
5. Які спільні риси мають РНК і ДНК-полімерази? У чому полягають принципові відмінності між ними?
6. Як здійснюється редагування помилок при синтезі ДНК?
7. Що таке реплісома? Назвіть її основні компоненти та їхнє функціональне значення.
8. Дайте визначення гелікази. Яку роль вона відіграє в реплікації? Що таке білки SSB?
9. Що таке праймер? Навіщо він потрібен, яка його хімічна природа, який елемент реплісоми його синтезує?
10. Яку роль виконує лігаза при реплікації ДНК?
11. Як забезпечується висока процесивність ДНК-полімерази?
12. Які топологічні проблеми виникають під час реплікації та за допомогою чого вони розв’язуються?
13. Дайте визначення ориджина. Як саме здійснюється ініціація реплікації в бактерій?
14. Укажіть особливості еукаріотичної системи реплікації порівняно з прокаріотичною.
15. Які ДНК-полімерази працюють в еукаріотичній клітині? Яка полімераза здійснює ініціацію реплікації?
16. Яку функціональну роль виконує теломераза? Опишіть механізм роботи цього ферменту.
17. Порівняйте ексцизійну репарацію основ і нуклеотидів.
18. Як здійснюється репарація місметчів у ДНК?
19. Яким чином відбувається репарація дволанцюгових розривів ДНК?
РЕКОМЕНДОВАНА ЛІТЕРАТУРА
1. Agarwal, S., Tafel, A.A., Kanaar, R. DNA doublestrand break repairand chromosome translocations // DNA Repair (Amst). – 2006. – Vol. 5. – Р. 1075 – 1081.
2. Baker, T.A., Bell, S.P. Polymerases and the replisome: machines withinmachines // Cell. – 1998. – Vol. 92. – Р. 295 – 305.
3. Demeret, C., Vassetzky, Y., Méchali, M. Chromatin remodelling andDNA replication: from nucleosomes to loop domains // Oncogene. – 2001. – Vol. 20. – Р. 3086 – 3093.
4. Donmez, I., Patel, S.S. Mechanisms of a ring shaped helicase // Nucleic Acids Res. – 2006. – Vol. 34. – Р. 4216 – 4224.
5. Dutta, A., Bell, S.P. Initiation of DNA replication in eukaryotic cells// Annu. Rev. Cell Dev. Biol. – 1997. – Vol. 13. – Р. 293 – 332.
6. Friedberg, E., Walker, G., Siede, W. DNA repair and mutagenesis. Washington, DC: ASM Press, 1995.Hübscher, U., Maga, G., Spadari, S. Eukaryotic DNA polymerases // Annu. Rev. Biochem. – 2002. – Vol. 71. – Р. 133 – 163.
7. Joyce, C.M., Steitz, T.A. Function and structure relationships in DNApolymerases // Annu. Rev. Biochem. – 1994. – Vol. 63. – Р. 777 – 822.
8. Kornberg A., Baker T.A. DNA replication. – New York: W.H. Freemanand Company, 1992.Kuriyan, J., O’Donnell, M. Sliding clamps of DNA polymerases // J. Mol.Biol. – 1993. – Vol. 234. – Р. 915 – 925.
9. Latt, W.L., de, Jaspers, N.G.J., Hoeijmakers, J.H.J. Molecular mechanismof nucleotide excision repair // Genes Dev. – 1999. – Vol. 13. – Р. 768 – 785.
10. Lindahl, T., Wood, R.D. (1999) Quality control by DNA repair // Science. – Vol. 286. – Р. – 897 – 1905.
11. Lohman, T.M., Bjornson, K.P. Mechanisms of helicasecatalyzed DNAunwinding // Annu. Rev. Biochem. – 1996. – Vol. 65. – Р. 169 – 214.
12. O’Donnell, M. Replisome architecture and dynamics in Escherichia coli//J. Biol. Chem. – 2006. – Vol. 281. – Р. 10653 – 10656.
13. Stillman, B. Cell cycle control of DNA replication // Science. – 1996. – Vol. 274. – Р. 1659 – 1664.
14. Zakian, V.A. Telomeres: beginning to understand the end //Science. – 1995. – Vol. 270. – Р. 1601 – 1606.
 2017-11-01
2017-11-01 541
541