Ошибки в ДНК возникают в результате неточной работы всех трех различных систем: подбора оснований полимеразами (base selection), редакторской 3'-5'-экзонуклеазы (proofreading) и репарации неправильно спаренных оснований (mismatch correction). Все это называется "спонтанный мутагенез", так как ошибки ДНК-полимеразы присущи самому организму, а не индуцируются извне. Однако, как известно, частоту мутаций можно значительно повысить, если организм обработать каким-либо ДНК-тропным агентом, например химическим веществом, ионизирующим или ультрафиолетовым излучением. Этот эффект называется индуцированным мутагенезом, а его механизм зависит от природы повреждения азотистого основания в ДНК. Все дефекты в ДНК удобно разбить на два класса: модифицированные азотистые основания, не блокирующие работу ДНК-полимеразы; и летальные дефекты, блокирующие работу ДНК-полимеразы. В первом случае ДНК-полимераза узнает дефектное основание и ставит напротив него комплементарное звено (например, при окислении аминогруппы цитозина, комплементарного гуанину, образуется азотистое основание с кетогруппой в четвертом положении, комплементарное аденину, в результате возникает транзиция G → А). Подобного типа дефекты носят исключительно мутагенный характер, а мутагены, индуцирующие в ДНК такие дефекты, широко используются на практике (например, производные алкилирующих соединений типа М-метил-N'-нитpo-N-нитрозогуанидина, метилметансульфоната, бисульфита и многих других).
Более сложная ситуация возникает, если в ДНК индуцируются летальные дефекты, блокирующие ДНК-полимеразу, например апуриновые/апиримидиновые сайты (abasic site или АР-сайты), циклобутановые пиримидиновые димеры (Руг-Руг) и др. В этом случае в клетке возникает особое состояние, получившее название SOS. Ключевыми элементами SOS-системы являются белки RecA и LexA. RecA уже известен нам по процессу рекомбинации (вспомним, что RecA - ключевой белок процесса гомологичной рекомбинации, способствующий формированию особого R-триплекса с гомологичным участком ДНК и переносу нити ДНК с одной молекулы ДНК на другую). LexA - репрессор группы генов (их количество около 30-40), входящих в состав SOS-регулона. LexA связывается со специфическим десятичленным палиндромом в регуляторных областях генов SOS-регулона и таким образом препятствует их транскрипции. Чем выше совершенство каждого данного палиндрома, тем крепче связан с ним репрессор LexA.
Белок RecA выполняет несколько функций. Во-первых, он имеет высокое сродство к однонитевой ДНК, поэтому при взаимодействии с онДНК образуется растянутый филамент RecA: ДНК (один мономер RecA на каждые 4 н. ДНК). Во-вторых, RecA в этом комплексе активируется (активированный RecA обозначают обычно RecA*) и приобретает свойства весьма специфической протеазы, деградирующей белок-репрессор LexA (точнее, ускоряющей авторасщепление белка LexА, то есть он может быть назван его копротеазой). В результате разрушения LexA открываются гены SOS-регулона, из которых два гена, umuD и umuС, имеют прямое отношение к индуцированному мутагенезу.
Здесь же отметим специфическую активность RecA* по отношению к белку UmuD. При взаимодействии RecA* с UmuD с N-конца UmuD отщепляются первые 24 аминокислотных остатка и образуется белок UmuD', который принимает непосредственное участие в SOS-мутагенезе.
Итак, в SOS-индуцированных клетках Е. coli в области повреждения формируется особая структура ДНК: вплоть до дефектного нуклеотида расположена двунитевая ДНК, с З'-ОН-концом последнего правильного нуклеотида которой связана ДНК-полимераза III, затем идут дефектный нуклеотид в матричной нити и однонитевая ДНК в виде филамента (RecA*: ДНК). Есть еще два уже упомянутых белка - UmuD' и UmuC, которые образуют комплекс (UmuD')2UmuC. Основная задача этого комплекса - "проскочить" сложный дефект и, желательно, без потерь, то есть без формирования бреши напротив дефекта. Подобный вариант синтеза и носит наименование TLS (translesion synthesis). Как правило, TLS сопровождается включением во вновь синтезируемую нить нуклеотида, некомплементарного исходному основанию в матрице. В результате возрастает выживаемость клетки (SOS-репарация) и возникает мутация (SOS-мутагенез). Этот процесс схематически показан на рис. 19 (Сойфер, 1997).
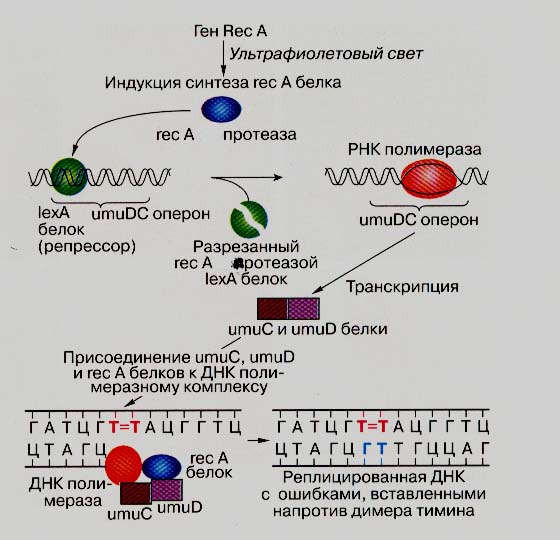
Рисунок 19. SOS-ответ у Escherihia coli.
При реконструкции этого процесса в системе in vitro было показано, что очищенный комплекс (UmuD')2UmuC в присутствии белков RecA, ATP и всех четырех dNTP самостоятельно ведет синтез ДНК, причем напротив G (перед дефектом) ставит С (dCTP, комплементарный синтез), а напротив X ставит случайный нуклеотид, то есть что этот комплекс является самостоятельной ДНК-полимеразой типа "error-prone", названной ДНК-полимераза V. Эффективность TLS (но не комплементарного синтеза) принципиально зависит от белка RecA и значительно усиливается при добавлении в смесь белка, связывающего однонитевую ДНК (SSB), и вспомогательных субъединиц ДНК-полимеразы III. В комплексе (UmuD')2UmuC собственно полимеразной активностью обладает UmuC, а субъединица (UmuD')2 является ее активатором. Показано, что механизм SOS-мутагенеза позволяет клетке заменить мутацию со сдвигом рамки (инсерция или делеция нуклеотида), как правило, инактивирующую ген, на более "мягкую" по последствиям мутацию замещения (транзицию или трансверсию).
В 1999 г. получены новые данные и по механизму мутагенеза на неповрежденной матрице ("non-targeted''-мутагенез). Известно, что "non-targeted''-мутагенез наблюдается при введении в SOS-индуцированные клетки неповрежденной матрицы. Показано, что белок DinB (ген dinB относится к группе SOS-генов) отвечает за "non-targeted''-мутагенез на ДНК бактериофага λ, а сверхпродукция этого белка приводит к появлению у клетки мутаторного фенотипа. Белок DinB также обладает полимеразной активностью. Он получил наименование ДНК-полимераза IV: эта полимераза не содержит редакторской экзонуклеазной активности и обладает гомологией с белками семейства UmuC. Таким образом, в клетках Е. coli к настоящему время обнаружены пять ДНК-полимераз: ДНК-полимераза I (репарирующая) обнаружена А. Корнбергом в 1956-1958 годах, ДНК-полимеразы II и III (реплицирующая) выявлены в 1970-1971 годах, а ДНК-полимеразы IV и V- в 1999 году. Долгое время оставалась непонятной роль ДНК-полимеразы II (кодирующий ее ген polB/dinA также относится к группе SOS-генов). Недавно было показано, что ДНК-полимераза II играет ключевую роль в ресинтезе ДНК. Известно, что при УФ-облучении бактерий репликация хромосомы блокируется, а затем через определенное время восстанавливается. Процесс восстановления репликации и называется ресинтезом, или "replication restart". Блокирование репликации определяется двумя факторами: SOS-индуцией umuDC (именно UmuD, но не UmuD'), которые на первой стадии действуют как регуляторы чекпойнтов клеточного цикла (подробнее о чекпойнт-контроле мы будем говорить ниже), увеличивая тем самым время, необходимое для более полного проведения эксцизионной репарации ДНК; и комплексом (UmuD')2UmuC, участвующим в процессе пострепликативной репарации ДНК. Каким именно образом ДНК-полимераза II восстанавливает синтез бактериальной хромосомы, остается пока неясным.
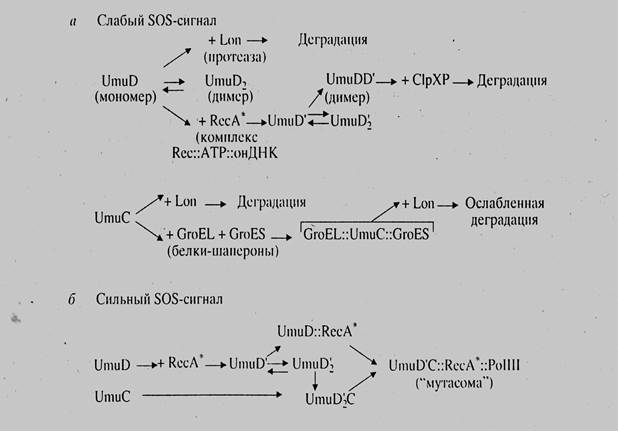
Рисунок 20. Роль интенсивности SOS-сигнала в процессе ответа клетки.
Помимо установления основных принципов действия SOS-системы in vitro, в последнее время получен ряд новых, принципиально важных результатов по регуляции SOS-системы in vivo. Оказалось, например, что белок UmuD расщепляется сериновой АТР-зависимой Lon-протеазой, а процессированный белок UmuD', у которого удалены с N-конца 24 аминокислотных остатка, в том числе фрагмент 15-FPLF, являющийся мишенью для Lon-протеазы, расщепляется (в комплексе с геликазой UvrD) другой клеточной сериновой АТР-зависимой протеазой ClpXP. К тому же белки RecA и LexA также являются членами SOS-регулона. В результате после SOS-индукции в бактериальной клетке довольно быстро восстанавливается уровень белков SOS-peгулона, характерный для необработанных клеток.
Интенсивность SOS-ответа клетки зависит от степени повреждения ДНК, так как только большое количество однонитевой ДНК позволяет активироваться RecA белку в количестве, достаточном для открытия SOS-регулона. Эта зависимость представлена на рис. 20 (Ланцов, 1999). Так как белок RecA связывается c LexA в том же сайте, в котором происходит его связывание с двунитевой ДНК, то при запуске SOS-ответа белок RecA не активирует процесс гомологической рекомбинации. В норме при гомологичной рекомбинации SOS-ответ также выражен слабо.
Каким же образом RecA* делает выбор между запуском SOS-ответа и инициацией гомологичной рекомбинации? Вероятно, все зависит от ситуации в клетке: быстрая диссоциация белка SSB от онДНК, то есть быстрое формирование комплекса белка RecA с онДНК приведет к SOS-ответу, который индуцирует также и синтез ряда рекомбинационных ферментов. Медленное формирование тройного комплекса завершается только гомологичной рекомбинацией со слабой SOS-индукцией. Скорость сборки комплекса зависит и от рекомбиногенной активности самого белка RecA.
На рис. 20 ясно видно, каким образом выраженность SOS-ответа зависит от количества повреждений ДНК.
8.1. Низкопроцессивные ДНК-полимеразы эукариот
У эукариот также обнаружены полимеразы, способные вести синтез на поврежденной матрице. Некоторые из них принимают самое активное участие в пострепликативной репарации, о которой мы будем говорить ниже. Номенклатура этих ДНК-полимераз была крайне не упорядочена до последнего времени. Только в 2001 году был достигнут некоторый консенсус, результаты которго представлены в табл. 5.
Например, полимераза эта (Polη) была описана разными авторами как polη, polΗ, hRad30, XPV. Эта полимераза не является малоточной, хотя и не обладает корректорской активностью. Точность ее репликации 10-2-10-3. Полимераза каппа (Polκ) является гомологом PolIV E.coli. Это высокоошибочная ДНК-полимераза, но в отличие от других подобных полимераз она может модулировать свою процессивность и встраивать больше, чем 25 нуклеотидов во время синтеза на поврежденной матрице. Полимераза йота (Polι) является гомологом Rad30 дрожжей и показывает уровень ошибочного встраивания 10-2. Полимераза зета (Polζ) состоит из двух субъединиц REV3 и REV7, которые работают совместно с REV1.
Кроме синтеза на поврежденной матрице многие из этих ДНК-полимераз способны к прямому устранению повреждения. Так, человеческая полимераза лямбда (Polλ) имеющая 32% гомологии с polβ, обладает лиазной активностью, способна вставлять нуклеотиды в маленькие бреши со свободной 5’-фосфатной группой и, вероятно, принимает участие в BER.
Меньше известно о полимеразе сигма (Polς, TRF4) (topoisomerase I related function), участвующей в когезии сестринских хроматид.
| Pol eta (Polη) | АА → (ТТ), А или G→ АР-сайта, А или С → 8-окси-G, C → сшивки |
| Pol kappa (Polκ) | A → АР-сайта или 8-окси-G |
| Pol iota (Polι) | A → АР-сайта или 8-окси-G, BER |
| Pol zeta (Polζ) | A или G → 6-4-фотопродуктов |
| Pol mu (Polμ) | A → АР-сайта или 8-окси-G, АА → (ТТ), V(D)J-рекомбинация, NHEJ |
| Pol lambda (Polλ) | BER |
| Pol sigma (Polσ) | Когезия сестринских хроматид |
| Pol theta (Polζθ) | Репарация межнитевых сшивок |
Таблица 5. Низкопроцессивные ДНК-полимеразы эукариот.
О полимеразе тета (Polθ) можно сказать только то, что она является гомологом гена mus308 дрозофилы, кодирующего ДНК-геликазу-полимеразу, вовлеченную в репарацию межнитевых сшивок.
Полимераза мю (Polμ) играет важную роль в соматическом гипермутагенезе иммуноглобулиновых генов и участвует в синтезе ДНК во время V(D)J рекомбинации и негомологического воссоединения концов. Человеческая Polμ также имеет высокую гомологию с терминальной дезоксинуклеотид-трансферазой, то есть способна к присоединению нуклеотида к концу нити ДНК без матрицы.. Все это говорит о большой плейотропности функций данной группы энзимов. В табл. 5 показано, какие нуклеотиды способна поставить данная полимераза напротив повреждения.
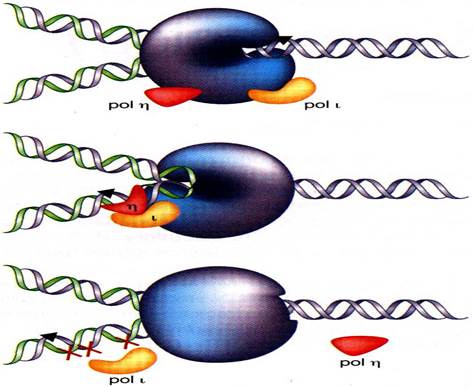
Рисунок 21. Синтез ДНК на поврежденной матрице.
Именно эта группа ДНК-полимераз ответственна за множественные мутации, возникающие при остановке репликации, вызванной повреждениями ДНК под действием различных агентов, и последующем ресинтезе (рестарте). Было несколько попыток связать деятельность этих полимераз с SOS функциями UmuDC у прокариот и, основываясь на этом, создать картину SOS–ответа у эукариот, Вопросов, к сожалению, до сих пор больше, чем ответов.
На рис. 21 изображена работа ДНК-полимеразы η, мутация в гене которой приводит к замещению ее полимеразой ι и развитию вариантной формы пигментной ксеродермы (XPV).
9. Репарация двунитевых разрывов.
Двунитевые разрывы ДНК (DSB, double strand breaks) являются наиболее тяжелыми повреждениями геномов эукариот и при отсутствии их репарации почти всегда приводят клетку к гибели. В частности, гибель клеток после действия ионизирующей радиации преимущественно связана с нерепарированными DSB, а также с хромосомными перестройками, возникающими при их неправильной репарации. DSB могут возникать спонтанно или после воздействия на клетку ионизирующей радиации и некоторых химических мутагенов.
В ядрах клеток эукариот находятся специфические для каждого вида наборы хромосом, причем одна хромосома содержит одну линейную молекулу ДНК. ДНК каждой хромосомы фланкирована особыми повторяющимися последовательностями, которые называются теломерами и составлены из большого числа тандемно расположенных блоков (TTAGGG)n. Между теломерами каждой хромосомы находятся экспрессирующиеся гены, «молчащие» последовательности ДНК, а также один особый небольшой эпигенетически заданный участок (центромера), необходимый для правильной сегрегации данной хромосомы в митозе, так как именно к нему прикрепляются нити веретена деления. Теломеры являются единственными «легально дозволенными» двунитевыми концами ДНК в клетке. Появление в молекуле ДНК нерепарированного DSB приводит к нарушению ее непрерывности и к образованию ацентрического хромосомного фрагмента, теряющегося при митозе. Если же процесс репарации DSB пройдет неточно и захватит не те концы, которые принадлежат одному и тому же разрыву, то в клетке появятся хромосомные перестройки, которые могут приводить к ее малигнизации. Каким образом это происходит, показано на рис. 22 (Томилин, 2002).
Одной из функций гомологической рекомбинации, которая, как мы уже говорили, возможна не только в мейотических, но и в соматических клетках позвоночных, является особый путь репарации двунитевых разрывов ДНК.
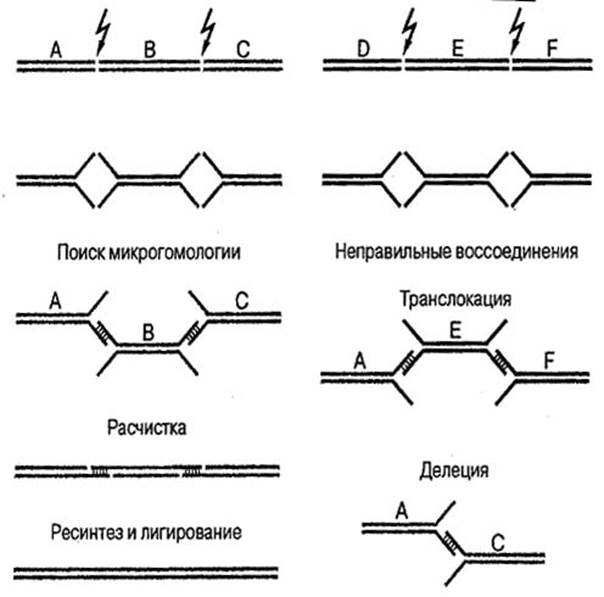
Рисунок 22. Воссоединение двунитевых разрывов ДНК.
Рекомбинационный механизм репарации DSB в дрожжах был впервые предложен в 1973 г. В. Г. Королевым и Л. М. Грачевой. Тогда предполагалось, что это - единственный возможный механизм репарации DSB, по своему генному контролю совпадающий с обычной гомологической рекомбинацией, однако оставалось неясным, работает ли такой механизм в соматических клетках позвоночных животных. Но к настоящему времени стало ясно, что рекомбинация не является основным способом репарации двунитевых разрывов. Репарация DSB путем гомологической рекомбинации (HRR, homologous recombination repair) происходит достаточно редко, так как для нее необходима вторая неповрежденная копия двунитевой ДНК, содержащая участок с нуклеотидной гомологией к поврежденному участку. При этом один из концов DSB внедряется в неповрежденную копию и инициирует обычный процесс генетической рекомбинации, так что понятия гомологической рекомбинации и рекомбинационной репарации у высших эукариот практически являются синонимами.
Главная опасность рекомбинационной репарации DSB в клетках позвоночных состоит в том, что их геномы содержат большое число повторов, по которым возможны «неправильные» гомологические взаимодействия, приводящие к хромосомным перестройкам за счет неравной рекомбинации. Неравная рекомбинация может и не приводить к хромосомным перестройкам, так как особенно эффективно гомологическая рекомбинация протекает путем генной конверсии между сестринскими хроматидами в поздней S-фазе и G2-фазе клеточного цикла. На механизмах, препятствующих неравной HR и появлению хромосомных перестроек между повторами мы остановимся отдельно.
Другими системами распознавания и репарации DSB в клетках эукариот являются прямое негомологическое воссоединение концов ДНК (NHEJ, nonhomologous end-joining) и отжиг по прямым повторам (SSA, single strand annealing). С них мы и начнем изучение репарации двунитевых разрывов.
 2015-05-10
2015-05-10 4404
4404